| December 3, 2024
Questo è un post riciclato dal periodo COVID e propone un’analisi esplorativa di un dataset COVID. L’intento è quello di mostrare come utilizzare R e un’analisi grafica statica per esplorare i dati. In casi come questo con svariate serie è spesso più utile utilizzare Plotly per costruire grafici interattivi.
Cosa si fa oggi
Oggi impariamo (velocemente) ad importare i dati e a fare dei grafici con R. Gli esempi che prendiamo in considerazione sono i dati che vanno di più negli ultimi mesi, cioé i dati su COVID 19. In particolare, ci concentriamo sui dati che sono divulgati dalla protezione civile.
Attenzione
In alto a destra nei riquadri con il codice c’è un’icona, cliccando sull’icona si copia tutto il codice del riquadro, che può essere inserito in R per farlo girare1 .
Importare i dati
Per importare i dati ci avvaliamo della funzione standard read.csv e dei suoi “wrapper”. Importiamo i dati direttamente dal sito github della protezione civile senza scaricarli prima.
datiNazionali = read.csv('https://raw.githubusercontent.com/pcm-dpc/COVID-19/master/dati-andamento-nazionale/dpc-covid19-ita-andamento-nazionale.csv')
sapply(datiNazionali, class)## data stato
## "character" "character"
## ricoverati_con_sintomi terapia_intensiva
## "integer" "integer"
## totale_ospedalizzati isolamento_domiciliare
## "integer" "integer"
## totale_positivi variazione_totale_positivi
## "integer" "integer"
## nuovi_positivi dimessi_guariti
## "integer" "integer"
## deceduti casi_da_sospetto_diagnostico
## "integer" "integer"
## casi_da_screening totale_casi
## "integer" "integer"
## tamponi casi_testati
## "integer" "integer"
## note ingressi_terapia_intensiva
## "character" "integer"
## note_test note_casi
## "logical" "logical"
## totale_positivi_test_molecolare totale_positivi_test_antigenico_rapido
## "integer" "integer"
## tamponi_test_molecolare tamponi_test_antigenico_rapido
## "integer" "integer"Con la seconda istruzione esaminiamo le classe delle variabili che compongono il dataframe importato, lo stesso risultato si sarebbe potuto ottenere con str(datiNazionali), ma con una visualizzazione diversa del risultato.
Notiamo che la funzione read.csv, ove possibile trasforma le variabili di testo in factor. Questo spesso è comodo ma in alcuni casi come nel caso della data non è adeguato, il comportamento può essere modificato con l’opzione as.is = TRUE; si consiglia di consultare l’aiuto della funzione per vedere anche le altre opzioni (?read.csv).
Ora diamo alla data un formato alla data un formato adeguato: poiché ci interessa solo la data eliminiamo la parte che non ci interessa in ogni osservazione e le trasformiamo in oggetti di classe data
datiNazionali$data <- strtrim(datiNazionali$data,10)
datiNazionali$data <- as.Date(strtrim(datiNazionali$data,10))Ora se guardiamo la classe della variabile vediamo
class(datiNazionali$data)## [1] "Date"ora abbiamo la variabile in un formato adatto ai nostri fini. Vi ricordo che in RStudio è possibile visualizzare la struttura degli oggetti di R anche dalla finestra environment.
In modo analogo possiamo importare i database a livello regionale e provinciale
datiRegioni= read.csv('https://raw.githubusercontent.com/pcm-dpc/COVID-19/master/dati-regioni/dpc-covid19-ita-regioni.csv')
datiProvince = read.csv('https://raw.githubusercontent.com/pcm-dpc/COVID-19/master/dati-province/dpc-covid19-ita-province.csv')
datiRegioni$data <- as.Date(strtrim(datiRegioni$data,10))
datiRegioni$codice_regione <- factor(datiRegioni$codice_regione)
datiProvince$data <- as.Date(strtrim(datiProvince$data,10))
datiProvince$codice_provincia = factor(datiProvince$codice_provincia)
datiProvince$codice_regione = factor(datiProvince$codice_regione)anche in questo caso ho convertito le date, notiamo che in questo caso lo ho fatto componendo le funzioni strtrim e as.Date senza fare le due trasformazioni fatte sopra. Inoltre, ho cambiato la classe delle variabili codice trasformandole in factor, mentre originariamente erano integer.
Rappresentazioni grafiche
A questo punto siamo pronti per un’analisi esplorativa dei dati, iniziamo dai dati a livello nazionale
plot(datiNazionali$data, datiNazionali$totale_casi, type = 'l', xlab = 'Data', ylab = 'Totale casi', main = 'Andamento nazionale totale casi', lwd = 1.5, col = 'darkred')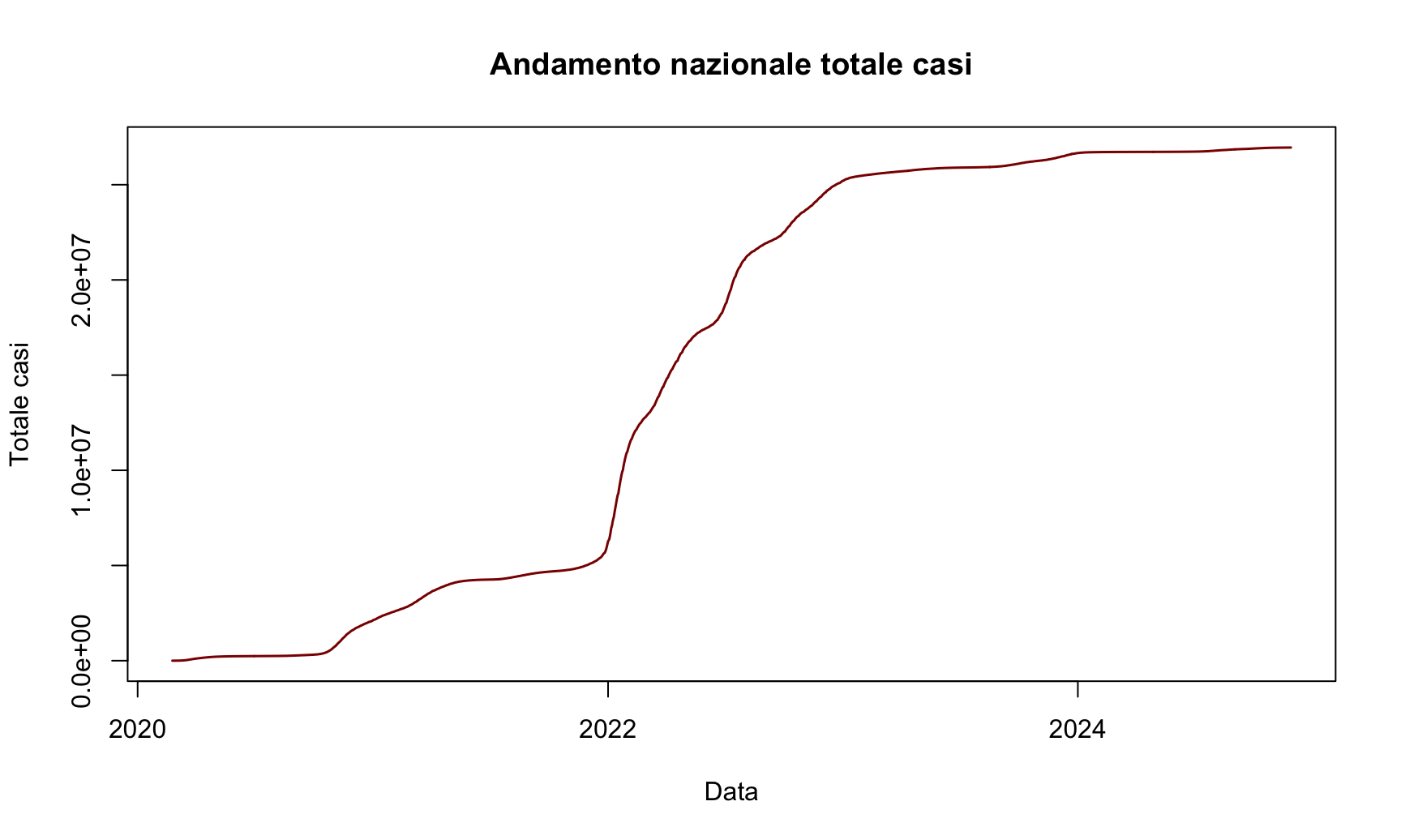 lo stesso risultato lo si può ottenere selezionando il dataframe contenente le variabili per l’ascissa e l’ordinata, come segue
lo stesso risultato lo si può ottenere selezionando il dataframe contenente le variabili per l’ascissa e l’ordinata, come segue
plot(datiNazionali[,c("data","totale_casi")], type = 'l', xlab = 'Data', ylab = 'Totale casi', main = 'Andamento nazionale totale casi', lwd = 1.5, col = 'darkred')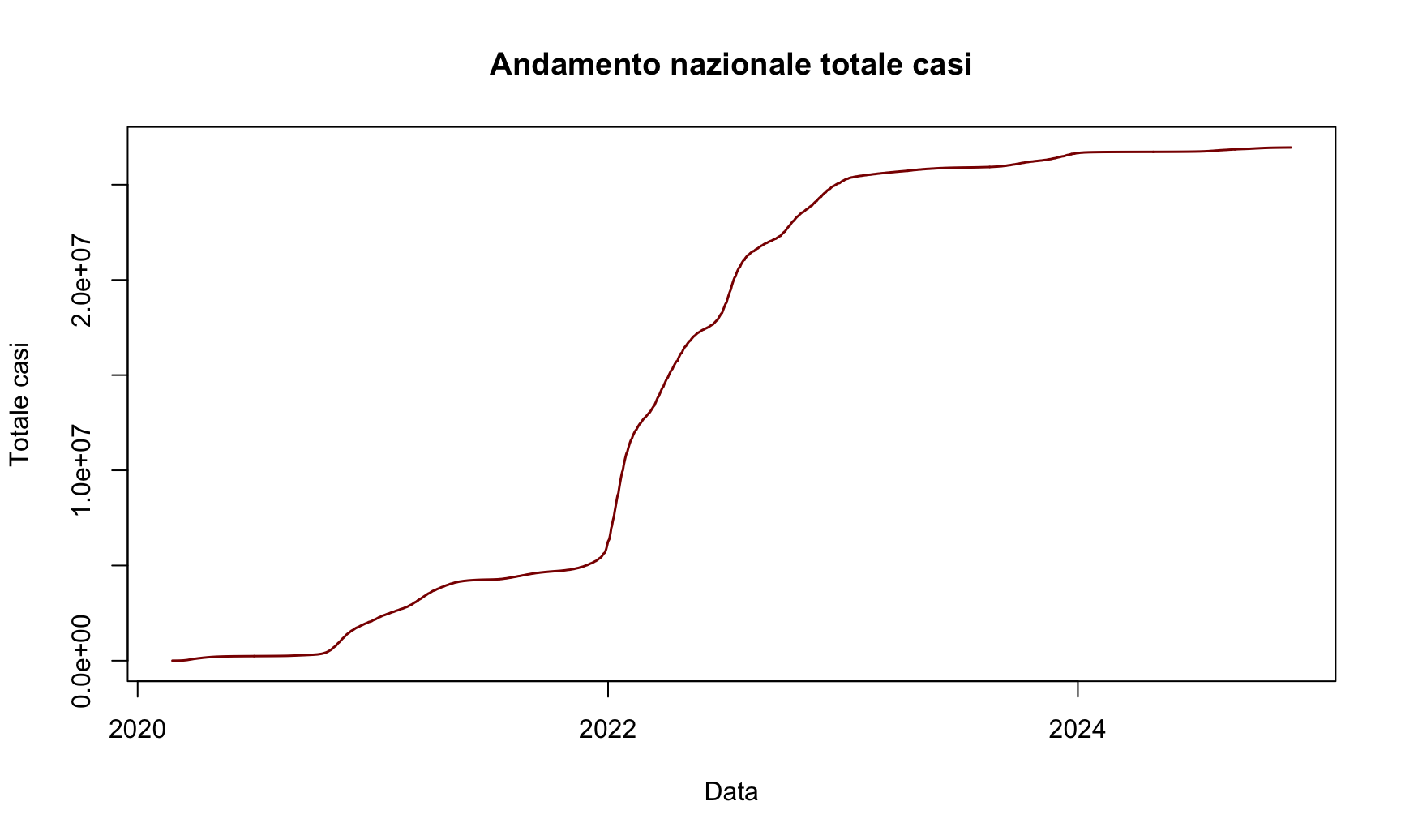
In modo simile possiamo rappresentare simultaneamente più variabili, ma per fare questo invece di utilizzare la funzione plot utilizziamo matplot:
vars = c('dimessi_guariti', 'deceduti', 'totale_ospedalizzati')
matplot(datiNazionali$data, datiNazionali[,vars], type = 'l', xlab = 'Data', ylab = 'Numerosità', main = 'Andamento nazionale', lwd = 1.5)
legend('topleft', vars, inset = .05, bty = 'n', lty = 1:3, col = 1:3, lwd = 1.5)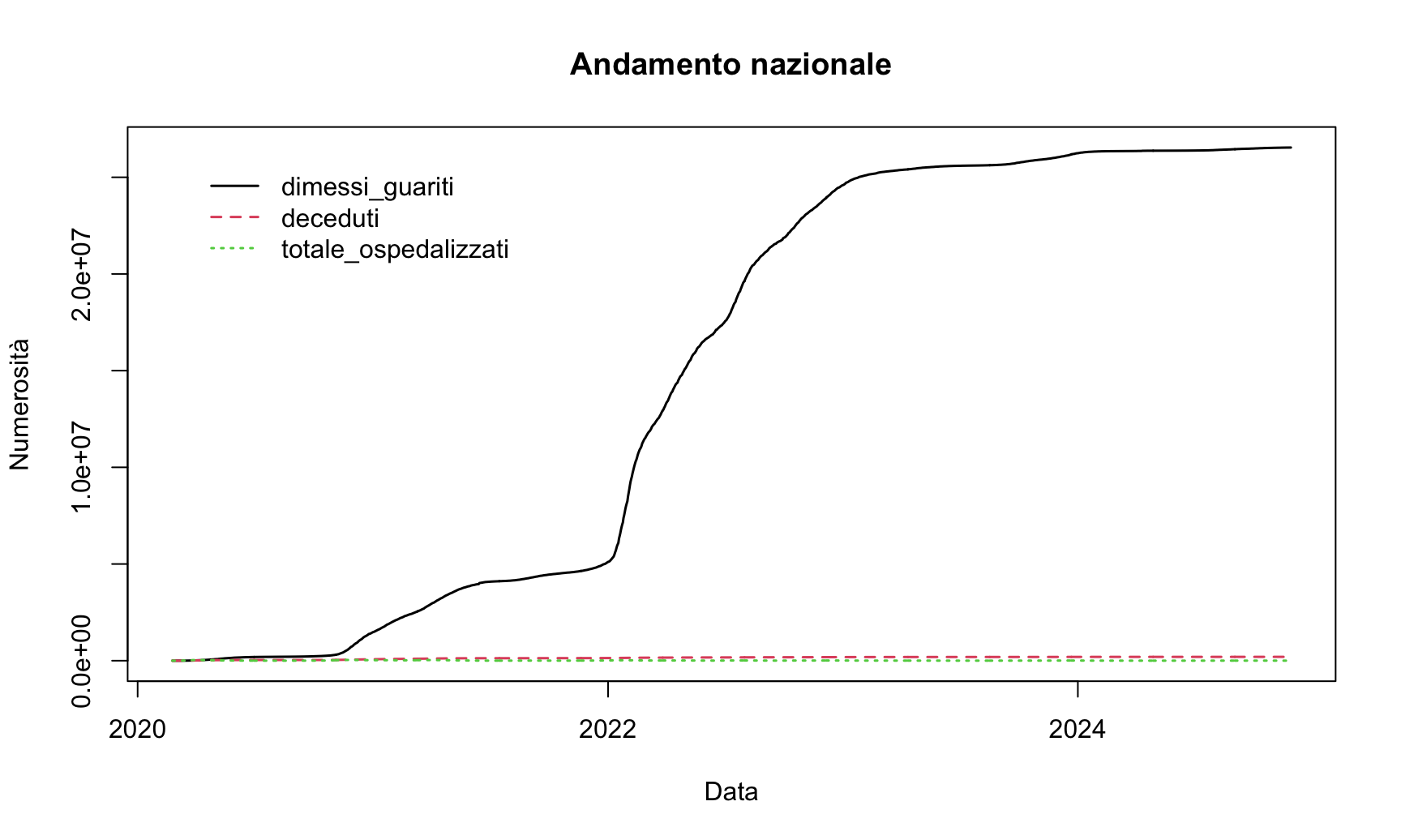
Con matplot le date però vengono visualizzate convertendole in valori numerici, il che non rende il grafico particolarmente leggibile, possiamo aggirare questo problema utilizzando la funzione plot come fatto in precedenza
vars = c('dimessi_guariti', 'deceduti', 'totale_ospedalizzati')
plot(datiNazionali[,c('data', vars[1])], type = 'l', xlab = 'Data', ylab = 'Numerosità', main = 'Andamento nazionale', lwd = 1.5)
foo = function(i) lines(datiNazionali$data, datiNazionali[,vars[i]], col = i, lty = i)
lapply(2:3, foo)## [[1]]
## NULL
##
## [[2]]
## NULLlegend('topleft', vars, inset = .05, bty = 'n', lty = 1:3, col = 1:3, lwd = 1.5)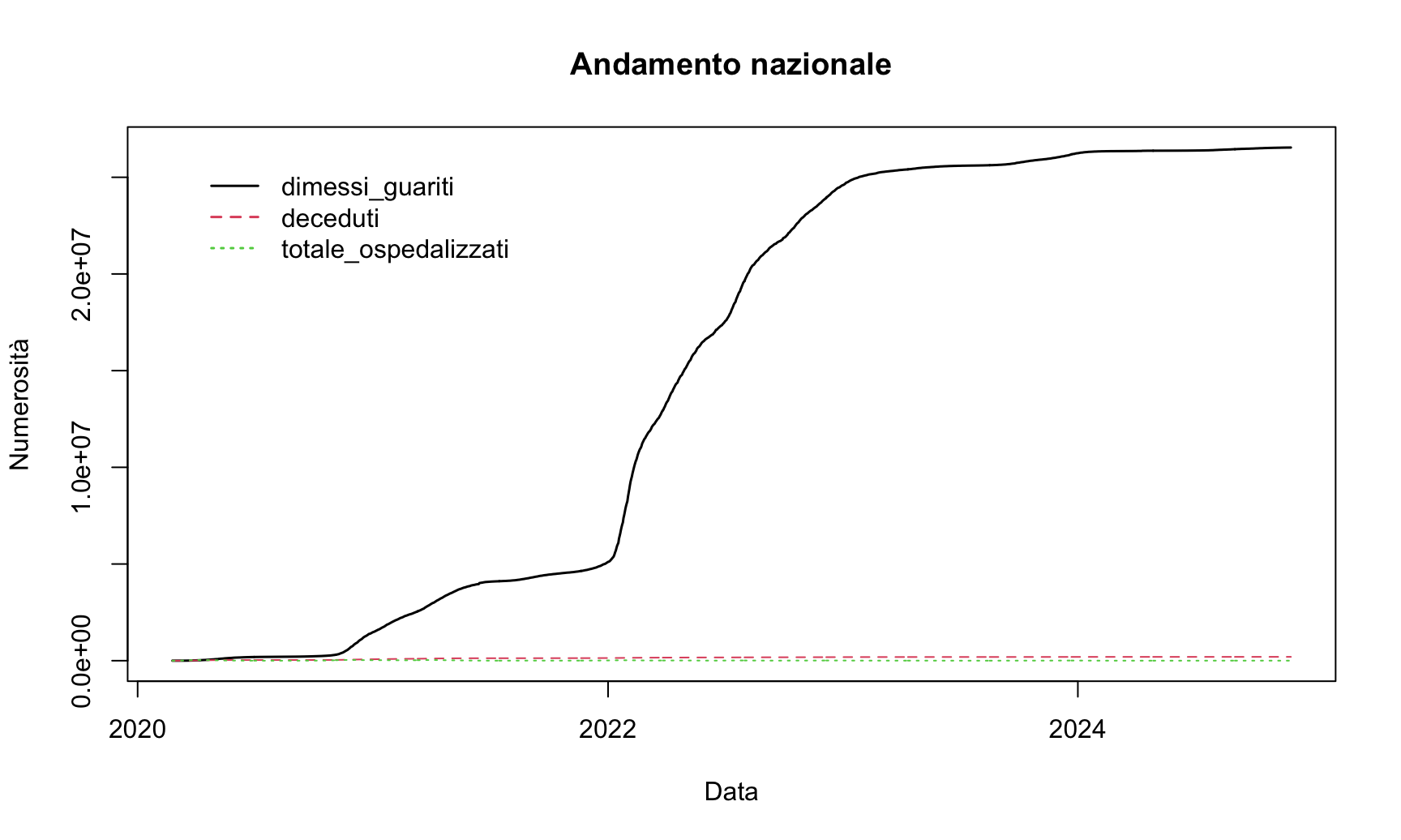
Notiamo che in questo caso ho inizializzato il grafico con la prima traccia, si sarebbe potuto fare anche a meno di fare la traccia, dopo di che ho aggiunto le altre tracce, per fare questo ho definito la funzione foo che calcolata in \(i\) aggiunge al grafico la linea della \(i\)-esima variabile della lista vars, quindi con lapply ho fatto aggiungere tutte le tracce mancanti. In questo modo variando il vettore vars possiamo ottenere il grafico che vogliamo, volendo possiamo definire direttamente una funzione che faccia questo per un generico vettore di variabili vars:
plotVars <- function(vars){
nVars = length(vars) ## numero variabili statistiche
yLim = c(0, max(datiNazionali[,vars]))
plot(datiNazionali[,c('data', vars[1])], type = 'l', xlab = 'Data', ylab = 'Numerosità', main = 'Andamento nazionale', lwd = 1.5, ylim = yLim)
foo = function(i) lines(datiNazionali$data, datiNazionali[,vars[i]], col = i, lty = i, lwd = 1.5)
lapply(2:nVars, foo)
legend('topleft', vars, inset = .05, bty = 'n', lty = 1:nVars, col = 1:nVars, lwd = 1.5)
}
vars = c("ricoverati_con_sintomi", "terapia_intensiva", "totale_ospedalizzati", "isolamento_domiciliare")
plotVars(vars)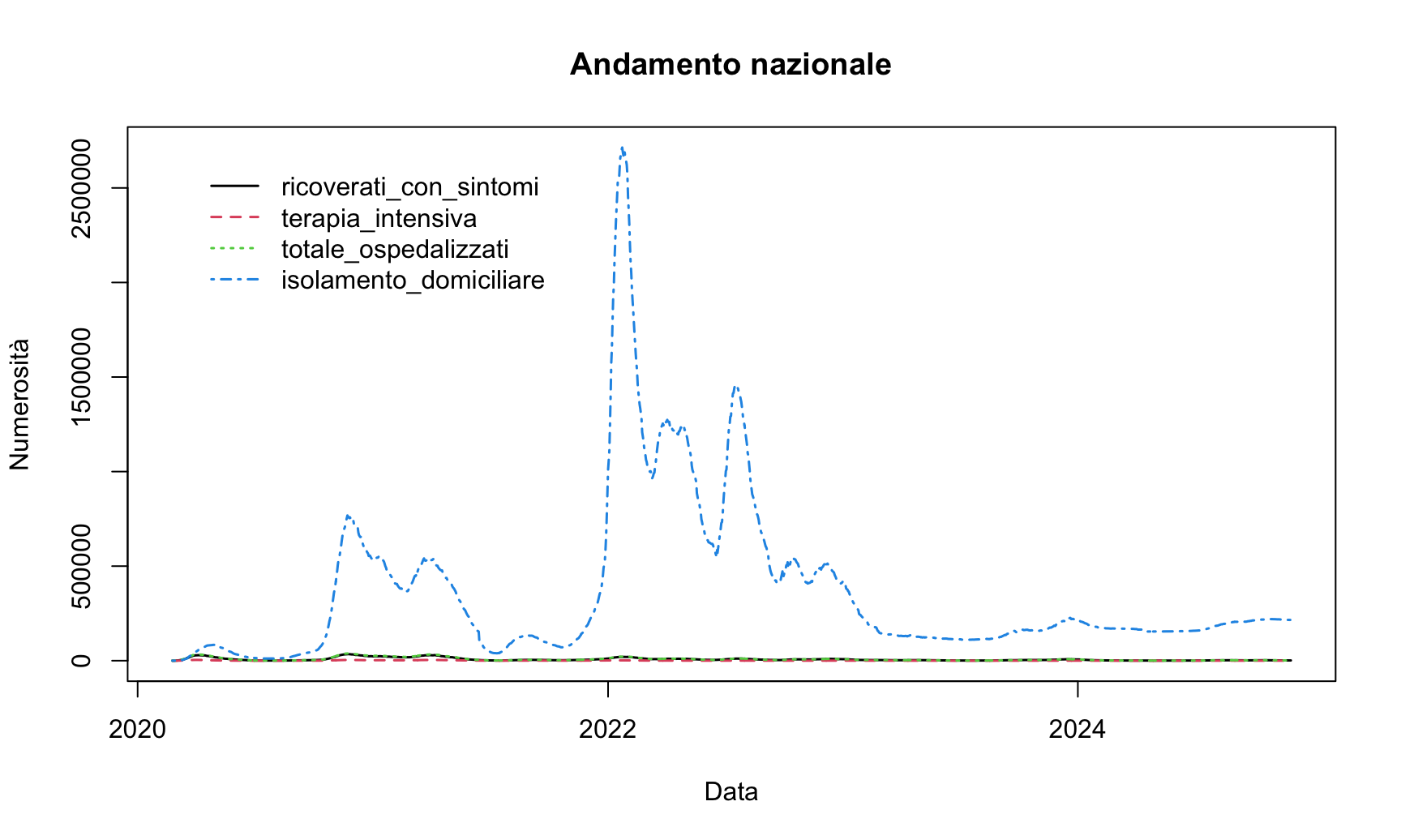
In questo caso abbiamo aggiunto al plot iniziale anche i limiti per l’asse delle ordinate, in modo tale che tutte le serie rientrino nei limiti del grafico, e la variabile nVar, che il numero di variabili che devono essere inserite nel grafico, questo perché il vettore vars ha una lunghezza non prestabilita.
Come andare avanti
Dopo questa prima analisi esplorativa dei dati a livello nazionale si può continuare l’esplorazione, in particolare studiando congiuntamente alcune variabili che ci possono dare particolari informazioni, ad esempio l’andamento del numero di tamponi e e il numero di casi positivi e così via.
Il passo successivo è passare allo studio dei dati regionali, infatti sappiamo che c’è una grande eterogeneità nella diffusione del virus a livello regionale, quindi è naturale confrontare il “trend” della pandemia nelle diverse regioni.
Notiamo che quando passiamo al dataset regionale le cose si complicano un po’ rispetto a quello nazionale, infatti la struttura è analoga a quello nazionale dal punto di vista delle variabili, ma è come si avessimo tanti dataset nazionali accostati per riga.
Il primo passo è “studiare” la nuova variabile regione, questa è rappresentata da due variabili, che sono
codice_regione: codice ISTAT associato alle regionidenominaazione_regione: nome esteso della regione
levels(datiRegioni$codice_regione)## [1] "1" "2" "3" "5" "6" "7" "8" "9" "10" "11" "12" "13" "14" "15" "16"
## [16] "17" "18" "19" "20" "21" "22"levels(datiRegioni$denominazione_regione)## NULLOsservando i livelli che possono assumere le variabili ci si accorge immediatamente che hanno un numero di livelli diversi, questo perché nella denominazione delle regioni il Trentino Alto Adige e suddiviso con le due province autonome, mentre nella classificazione con i codici vi è la regione nella sua interezza.
Dato che mi interessa solo la regione creo la serie sommando le osservazioni nelle due province autonome, il codice è il seguente:
temp = datiRegioni[datiRegioni$codice_regione == 4, c(1,7:18)]
temp = datiRegioni[, c(1,7:18)]
temp = aggregate(temp[,-1], by = list(temp$data), sum)
datiRegioni$denominazione_regione = factor(datiRegioni$denominazione_regione)
levels(datiRegioni$denominazione_regione)[13] = 'Trentino - Alto Adige'
datiRegioni = datiRegioni[datiRegioni$denominazione_regione != "P.A. Bolzano",]
datiRegioni[datiRegioni$codice_regione == 4, 7:17] = temp[,-1]
datiRegioni$denominazione_regione = factor(datiRegioni$denominazione_regione)
rm(temp)
levels(datiRegioni$denominazione_regione)## [1] "Abruzzo" "Basilicata" "Calabria"
## [4] "Campania" "Emilia-Romagna" "Friuli Venezia Giulia"
## [7] "Lazio" "Liguria" "Lombardia"
## [10] "Marche" "Molise" "Trentino - Alto Adige"
## [13] "Piemonte" "Puglia" "Sardegna"
## [16] "Sicilia" "Toscana" "Umbria"
## [19] "Valle d'Aosta" "Veneto"Il codice scritto sopra per fare questo non penso sia il più semplice, ma funziona, e ome si vede ora abbiamo le venti regioni italiane.
A questo punto possiamo fare una prima rappresentazione grafica
plot(datiRegioni$data, datiRegioni$totale_casi, pch = 20, col = rainbow(20)[datiRegioni$codice_regione], xlab = 'Data', ylab = 'Numero casi')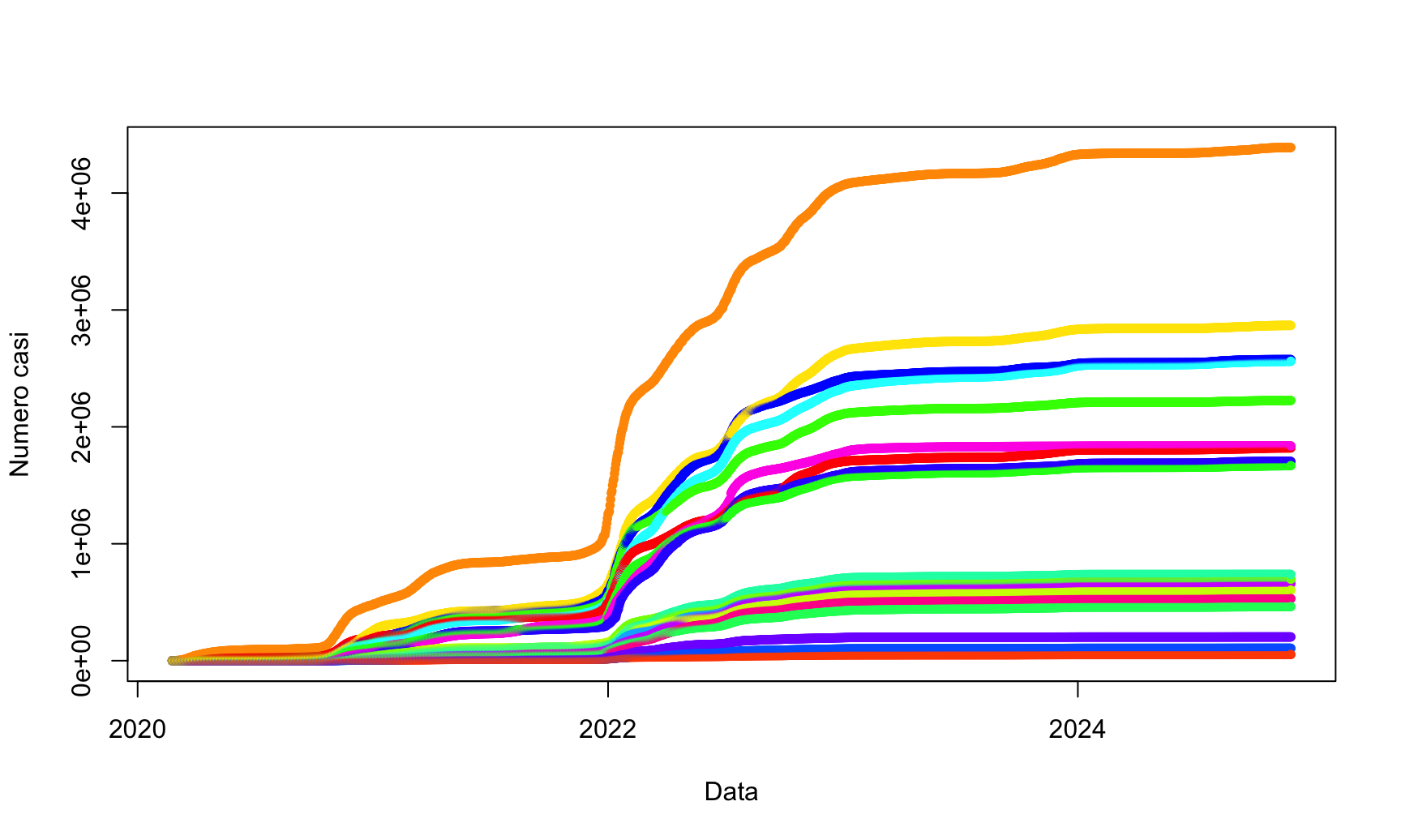 A questo punto si potrebbe anche aggiungere la legenda, ma ovviamente il grafico è di difficile lettura, quindi è meglio cambiare rappresentazione o passare a più grafici per descrivere lo stesso fenomeno. Questo lo vedremo un’altra volta.
A questo punto si potrebbe anche aggiungere la legenda, ma ovviamente il grafico è di difficile lettura, quindi è meglio cambiare rappresentazione o passare a più grafici per descrivere lo stesso fenomeno. Questo lo vedremo un’altra volta.
Come si può fare per produrre un grafico analogo al precedente, ma con le linee al posto dei punti? Posso usare la funzione plot come fatto in precedenza?
Per risolvere questo problema si consiglia di studiare la funzioni aggregate e split.